Bioinformática - Análise Filogenética com Clustalx
O Clustalx é um software para o alinhamento de sequências nucleotídicas, peptídicas e analises filogênicas. O Clustalx é software semi-livre segundo a sua forma de licenciamento. Embora possua características de software livre como permissão de execução de programa, não há permissão de uso para fins comerciais.
[ Hits: 29.234 ]
Por: José Cleydson Ferreira da Silva em 08/07/2010
Usando o Clustalx

- Gap Opening- penalização de escore para iniciar uma região de "Gap".
- Gap Extension- penalização de escore para estender uma região de "Gap" (normalmente menor que o Gap Opening)
- Delay Divergent Sequences - atraso de alinhamento de sequências divergentes que somente serão alinhadas após as outras sequências (porcentagem de identidade abaixo da qual a sequência será considerada divergente)
- Transition Weight (somente DNA) - da a transições (A<->G, T<->C) um escore diferente de 0.
- Use negative matrix - permite o uso de matrizes negativas, importante quando as sequências forem relacionadas somente em uma pequena porção. Em condições normais prejudica um pouco o alinhamento.
- Protein Weight Matrix - matriz de substituição a ser utilizada. Note que você só escolhe a serie de matriz: Blosum, PAM etc, o tipo de matriz dentro desta série (por exemplo Blosum 62, blosum 80) é escolhido automaticamente pelo programa.
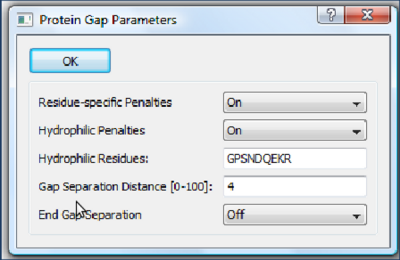
- Residue-specific Penalties - considera a vizinhança de alguns resíduos como mais ou menos favorável para a abertura de "Gaps".
- Hydrophilic Penalties - aumenta a chance de Gaps em regiões ricas em resíduos hidrofílicos que, usualmente, representam regiões menos estruturadas.
- Hydrophilic Residues - especifica quais resíduos são considerados hidrofílicos.
- Gap Separation Distance - número de resíduos de distância de uma região com Gap na qual é penalizada uma nova abertura de Gap.
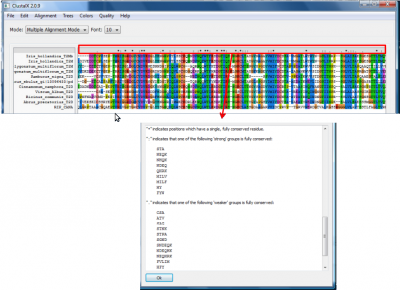
A apresentação de resultado de um alinhamento no ClustalX é representada por símbolos que indicam a conservação e os resíduos ou grupos de resíduos em uma coluna.
- Low Scoring Segments - mostra, em cinza, regiões com baixo escore e que, portanto, não seriam muito confiáveis
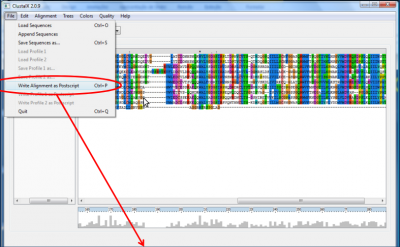
Também é possível exportar o alinhamento em formato Postscript através dos atalhos Ctrl+P ou acionando o menu File opção Write Alignment as Postscript. A visualização do arquivo pode ser feita por meio de um visualizador de imagens.
2. Usando o Clustalx
3. Sobre o autor
Bioinformática - PhyML: alinhamento de sequências nucleotídicas em ambiente paralelo
Cairo-Dock - Seu desktop Linux com cara de MAC
Novidades e mudanças na estrutura e configuração do Grub2, fique por dentro!
Bioinformática - Instalação do Mr Bayes em ambiente paralelo
Acessando desktop remotamente com noMachine
Asterisk: Entendendo o arquivo extensions.conf
Instalação de template para monitoramento de servidor Squid e servidor LDAP no CACTI (Debian)
Um tour pelos players de áudio para Linux
Nenhum comentário foi encontrado.
Patrocínio
Destaques
Artigos
WhatsApp com Chamadas no Linux via Waydroid
XFCE - quase um Gnome ou Plasma mas muito mais leve
LXQT - funcional para máquinas pererecas e usuários menos exigentes
Dicas
Conheça o Zashterminal, um terminal moderno com IA
DOOM Carniceiro: rode o Meatgrinder com uzdoom (Gentoo e Ubuntu)
Samba 4 AD-DC 2026: Como instalar e configurar um Active Directory (via APT-GET)
[Resolvido] Sumiço de redes e micro quedas no iwd/iwgtk (Realtek rtw88)
Como Configurar DNS Reverso (PTR) em Ambientes Linux e Microsoft
Tópicos
Linux MInt nao reconhece placa de som (2)
Midia de instalação LM-21.3 não inicializa (12)
Top 10 do mês
-

Xerxes
1° lugar - 118.634 pts -

Fábio Berbert de Paula
2° lugar - 56.215 pts -

Buckminster
3° lugar - 25.344 pts -

Mauricio Ferrari (LinuxProativo)
4° lugar - 18.385 pts -
Alberto Federman Neto.
5° lugar - 17.444 pts -

Sidnei Serra
6° lugar - 17.372 pts -

Alessandro de Oliveira Faria (A.K.A. CABELO)
7° lugar - 17.337 pts -

Daniel Lara Souza
8° lugar - 17.099 pts -

edps
9° lugar - 16.750 pts -

Diego Mendes Rodrigues
10° lugar - 16.557 pts